Il nostro Centro di Imaging sperimentale svela il comportamento del "guardiano del DNA"
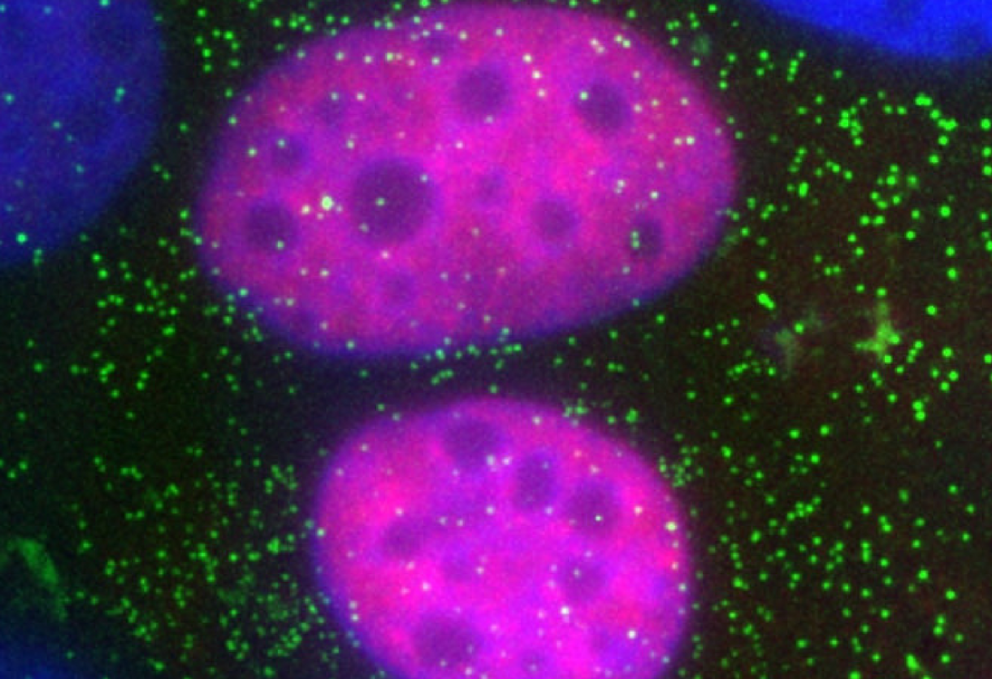
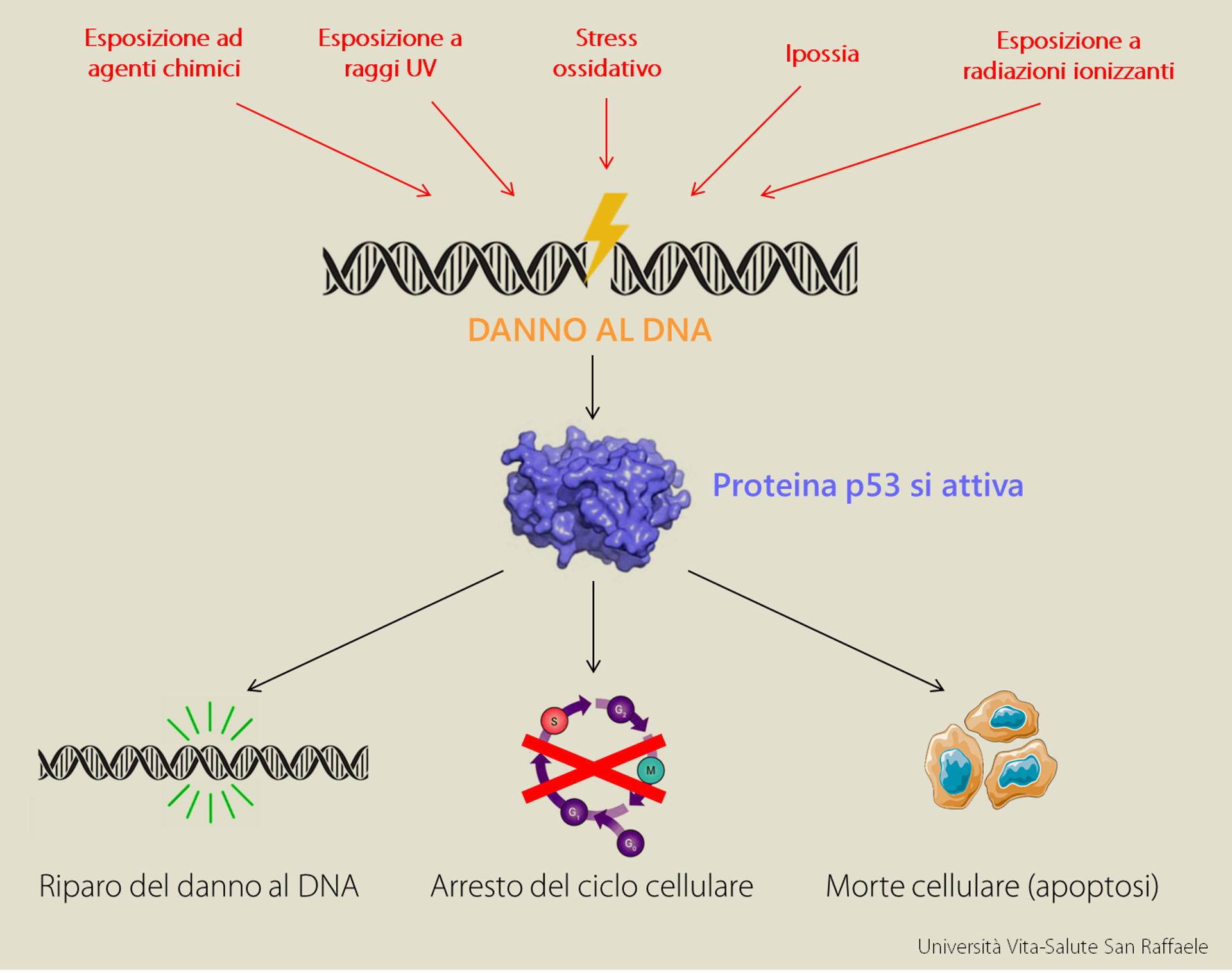
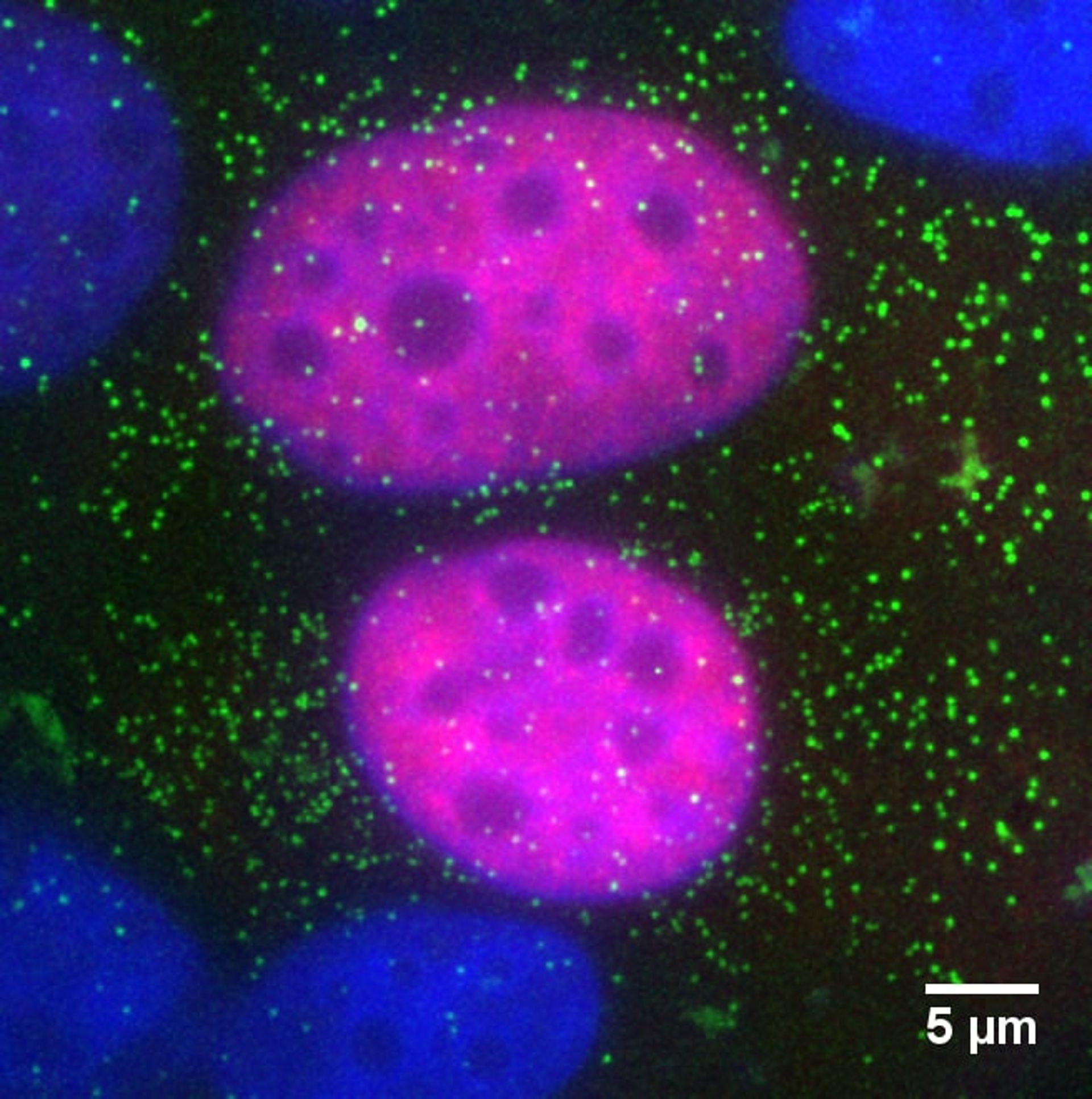
Quando il DNA di una cellula viene danneggiato, una proteina detta p53 si attiva per prendere una decisione fondamentale: avviare il processo di riparazione del DNA o indurre la morte cellulare. Il suo obiettivo, in entrambi i casi, è quello di evitare che la cellula diventi tumorale, un ruolo talmente decisivo da aver fatto guadagnare a tale proteina il soprannome di “guardiano del genoma”.
Per questa intervista abbiamo incontrato Davide Mazza, Alessia Loffreda e Carlo Tacchetti, autori di un articolo pubblicato sulla prestigiosa rivista Nature Communications, che – grazie a una sofisticata tecnologia di microscopia – hanno osservato per primi in tempo reale la dinamica di p53 nel nucleo delle cellule e ne hanno svelato il meccanismo di attivazione.
COS’È P53 E PERCHÉ È COSÌ IMPORTANTE PER LE CELLULE
“La proteina che studiamo si chiama p53, cruciale nella risposta al danno cellulare. Il suo ruolo è quello di “tradurre” il segnale di danno al DNA in una risposta cellulare che può portare la cellula a risolvere o riparare il danno, altrimenti, se il danno è irreparabile, alla morte cellulare (apoptosi). Questa risposta mediata da p53 è importantissima per evitare la progressione della cellula normale in cellula tumorale; non a caso circa il 70% dei tumori presenta una mutazione nel gene che la produce” spiega il Dott. Davide Mazza, fisico e Project Leader del Centro di Imaging dei Tumori. “Il primo evento che si osserva a seguito di un danno cellulare è un aumento dei livelli di espressione di p53. Per lungo tempo si è creduto che questo aumento fosse sufficiente a indurre una risposta al danno, ma negli ultimi tempi in letteratura sono emersi dati contrastanti a riguardo”.
La loro ricerca si inserisce proprio in questo punto: “Io sono un fisico, mi sono sempre occupato di microscopia, e a cavallo tra il mio postdoc negli Stati Uniti e l’inizio della mia attività di ricerca al San Raffaele ho messo a punto una tecnica innovativa che permette di misurare il comportamento dinamico di qualsiasi proteina cellulare a livello di singola molecola: questo consente di seguire – molecola per molecola – come si comporta una proteina. Il mio interesse si è focalizzato su p53 perché è un target fondamentale e interessantissimo da studiare: grazie a questa tecnica avanzata di microscopia abbiamo potuto misurare il comportamento e l’associazione di p53 ad altre proteine e al DNA, scoprendo che la “decisione” di p53 dipende dalle modifiche che avvengono a suo carico”.

p53 è al centro di una struttura “a clessidra”: molteplici stimoli (esposizione ad agenti chimici, raggi UV, radiazioni, stress genotossico, ipossia…) devono essere decodificati in altrettante risposte (riparo del danno al DNA, arresto del ciclo cellulare, morte cellulare…). Come fa questa proteina a decodificare diversi stimoli in risposte diverse? Una possibilità è che, a seconda di come viene modulata, possa regolare in maniera differente il suo legame a opportuni siti sul DNA, modificando dunque l’espressione di geni che attiveranno le varie risposte.
“Una delle modificazioni fondamentali che abbiamo identificato avvenire a carico di p53 è la sua acetilazione al C terminale [vale a dire l’aggiunta del gruppo chimico “acetile” ad una delle due estremità, denominata “C”, della proteina, N.d.R.]. Tali modifiche influenzano in modo importante la durata degli eventi di legame, ad esempio definiscono il “tempo di residenza” di p53 sul DNA. Questo risultato è sorprendente, in quanto il nostro lavoro dimostra come – rispetto al semplice aumento dei livelli di espressione di p53 – simili eventi molecolari siano in grado di predire molto meglio il comportamento cinetico di p53”.
Esistono alcuni tumori che invece non presentano una versione mutata di p53, quali ad esempio il neuroblastoma, un tumore del cervello molto difficile da diagnosticare in tempo per il trattamento, e che colpisce soprattutto i bambini. In questi tumori p53 potrebbe essere inattivato proprio a causa del meccanismo d’azione scoperto dai ricercatori del San Raffaele: conoscere questi sistemi per poi agire su di essi sarà uno degli obiettivi degli scienziati del CIS.
IL CENTRO DI IMAGING SPERIMENTALE (CIS)
Interviene il Prof. Carlo Tacchetti, Direttore del Centro di Imaging Sperimentale (CIS) e Ordinario di Anatomia Umana presso la Facoltà di Medicina e Chirurgia del nostro Ateneo: “Il filone di ricerca che Davide sta seguendo nel nostro Centro è molto rilevante, e certamente non isolato; al contrario, il Centro di Imaging Sperimentale è inserito in un contesto di grande collaborazione, che si sta sempre più ampliando su molteplici fronti. Dal punto di vista tecnologico, il metodo ideato da Davide di quantificare a livello di singola molecola l’affinità tra una proteina nucleare e i suoi siti di legame sul DNA, sta destando sempre maggior interesse, tanto da permetterci di instaurare una serie di collaborazioni con molti gruppi internazionali (tendenzialmente in campo oncologico)”. Tra i gruppi in cooperazione con il CIS ci sono, ad esempio, Michele Carbone alla University of Hawaii, Colin Goding al Ludwig Institute for Cancer Research in Oxford, Edouard Bertrand al CNRS di Montpellier, Pier Paolo di Fiore all’IFOM di Milano. “Nell’allestire il CIS abbiamo seguito la filosofia secondo la quale l’innovazione tecnologica consente la soluzione di problemi scientifici, generando così nuove domande aperte su cui lavorare, in una continua e stimolante esplorazione dei più svariati ambiti scientifici. Inoltre – specifica il Prof. Tacchetti – il punto di forza del CIS è la sua miscela opportunamente dosata di molteplici competenze: dai fisici agli ingegneri informatici, dai medici ai biologi, ciascuno mette a disposizione entusiasmo, capacità e preparazione professionale. Una ‘ricetta’ che si sta rivelando vincente, tanto da posizionarci come il centro di imaging attualmente più avanzato l’Europa a livello omnicomprensivo”.

Conclude la Dott.ssa Alessia Loffreda, postdoc nel laboratorio del Dott. Mazza e prima autrice dello studio: “Inizialmente l’analisi dei filmati ottenuti con questo innovativo microscopio veniva fatta quasi tutta a mano: oltre ad essere incredibilmente laboriosa, introduceva anche un serio pericolo di bias [errore, distorsione del dato, N.d.R.]. Il sistema è oggi invece completamente automatizzato, grazie alle competenze tecniche di Davide e quelle biologiche e biotecnologiche dei membri del nostro team. Sono molto contenta di essere primo autore di questo studio: si tratta di una tappa considerevole in ambito biomedico, che rende il CIS ancora più sicuro riferimento cui istituzioni nazionali e internazionali possono affidarsi”.
Lo studio è stato possibile grazie al finanziamento dell’IRCCS San Raffaele di parte della strumentazione necessaria alla costruzione del prototipo di microscopio sviluppato per questo studio e messo poi a disposizione della comunità scientifica dell’Istituto, ad un finanziamento Marie Curie International Incoming Fellowship, e ad un finanziamento da parte della Fondazione Cariplo relativo al “Bando Ricerca biomedica condotta da giovani ricercatori”, vinto anche grazie a una collaborazione con il Centro Europeo di Nanomedicina.
You might be interested in

World Rare Disease Day: Research at UniSR Continues All Year Round

Multiple sclerosis, breakthrough in research: researchers have identified a molecule that promotes repair of the nervous system
The microbiome as an ally against myeloma

